Введение
Рибонуклеиновая кислота (РНК) содержит генетическую информацию и используется организмом для кодирования конкретного интересующего белка. Результаты использования специфической разновидности РНК, а именно мРНК (матричная рибонуклеиновая кислота, или messenger RNA, – особый тип клеточных РНК, который содержит копию последовательности одного гена [1]) в качестве потенциальной технологии доставки генов in vivo были опубликованы в 1990 г., когда было показано, что прямая инъекция «свободной» мРНК приводила к экспрессии кодируемого белка in vivo [2]. Тем не менее ряд проблем препятствовал немедленному использованию in vitro транскрибированной мРНК в качестве простого средства для синтеза продукта in vivo, условно говоря, в виде простой инъекции. Эта проблема связана прежде всего с нестабильностью мРНК in vivo из-за почти повсеместного присутствия РНКаз. мРНК оказалась иммуногенной, стимулируя ответы с сопутствующим снижением трансляции мРНК. Прогрессом, который привел к быстрому наращиванию усилий по использованию мРНК в качестве платформы, было открытие K. Karikó и D. Weissman того факта, что использование модифицированных нуклеозидов приводит к снижению иммуностимулирующих эффектов транскрибируемой in vitro мРНК за счет уменьшения стимуляции Toll-подобных рецепторов [3]. Разработка средств доставки, в основном липосом/липонаносом, которые помогают стабилизировать мРНК и облегчают ее доставку в клетки и высвобождение из эндосом [4] (и, вероятно, действуют как адъюванты для кодируемого белка), также играет ключевую роль. Описание различных структурных вариантов липосом вы можете найти в ряде обзоров [5, 6].
Создание вакцин против SARS-CoV-2 (Severe acute respiratory syndrome-related coronavirus 2) на основе мРНК, кодирующей спайк (spike, S)-гликопротеин вируса – ключевой компонент связывания вируса с клеткой хозяина [7], стало действительно прорывом в вакцинопрофилактике и вирусологии в целом, принимая во внимание отсутствие зарегистрированных вакцин на основе мРНК до момента появления первых двух вакцин против SARS-CoV-2, созданных компаниями Pfizer/BioNTech и Moderna [8–10]. Вакцины указанных двух компаний стали первыми, зарегистрированными в США и ЕС вакцинами против SARS-CoV-2 с начальной заявленной эффективностью на уровне примерно 95% [9–11]. Общие данные в отношении существующих типов вакцин, в т.ч. и разрабатываемых в отношении SARS-CoV-2, можно найти в ряде литературных обзоров [12, 13].
Если кратко и предельно схематично описать механизм работы вакцин на основе мРНК [14], то следует обозначить несколько основных этапов: 1) создание молекулы мРНК, кодирующей S-гликопротеин SARS-CoV-2, и помещение ее в транспортную субстанцию (в нашем случае – липосому); 2) попадание липосомы в составе вакцины в организм человека; 3) слияние липосомы с клеткой, выход мРНК в цитоплазму, синтез рибосомами S-гликопротеина; 4) презентация антигенов – белковых компонентов S-гликопротеина или его отдельных структур на поверхности инфицированной клетки или специфических антиген-презентирующих клеток; 5) активация гуморального (синтез антител) и клеточного иммунитета (Т-клетки); 6) формирование иммунной памяти и ответ иммунной системы при последующей встрече с вирусом.
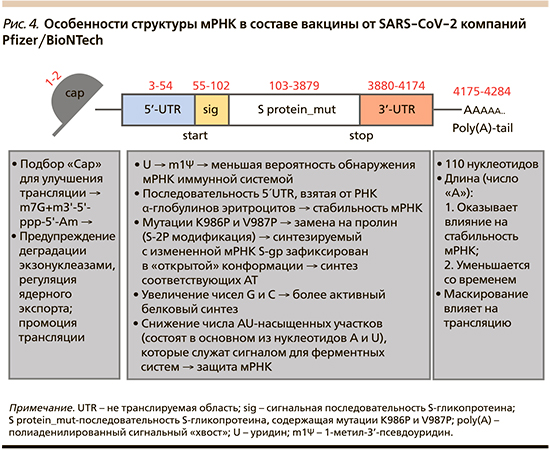
Молекула мРНК, в большей степени ее стабильность, т.е. время жизни, является краеугольным камнем эффективности вакцин, однако, как показывает практика, представления не только обывателей, но и научных работников и врачей идет по пути интуитивного упрощения, когда мРНК представляется стандартной структурной единицей, описание которой можно найти в любом учебнике по биологии или в сети Интернета, например в Википедии [15]. Структура молекулы мРНК, кодирующей S-гликопротеин SARS-CoV-2, представлена на рис. 1 и вполне очевидно визуально она не демонстрирует принципиальных различий с какой-либо другой мРНК. Однако не все так просто, и для стабилизации работы мРНК компаниям пришлось осуществить отбор среди ряда претендентов, внося ряд структурных изменений и оценивая их влияние на стабильность, иммуногенность и другие свойства продукта. В данной статье мы охарактеризуем структурные особенности мРНК в составе вакцины компании Pfizer/BioNTech (BNT162b2, или Tozinameran, или Comirnaty) [16, 17]. Это стало возможным благодаря тому, что в декабре 2020 г. на сайте Всемирной организации здравоохранения (ВОЗ) [18] была опубликована полная расшифровка генома мРНК в составе вакцины Pfizer/BioNTech, а затем на веб-сайте ученого и антрепренера Bert Hubert, основателя компании PowerDNS [19], была сделана попытка разобрать особенности структуры мРНК в составе вакцины указанной компании, и представляется практически полезным предпринять попытку воспроизвести наиболее интересные характеристики мРНК в составе вакцины Pfizer/BioNTech, что позволит нам четко понять, что процесс создания вакцин на основе мРНК – как минимум процесс создания инкорпорируемой молекулы рибонуклеиновой кислоты помимо других этапов синтеза и финализации продукта – отнюдь непростая задача.

Немецкая компания BioNTech, имея широкий спектр технологий in vitro транскрибированных (ivt) мРНК, с целью получения максимально безопасной и эффективной вакцины для профилактики SARS-CoV-2 в январе 2020 г. разработала 4 различных типа вакцин на основе ivt мРНК: две мРНК, кодирующие полноразмерный S-гликопротеин в «prefusion» конформации, одна из которых содержала в своем составе псевдоуридин, другая – немодифицированный уридин (на тот момент времени не было доказательств того, что для этого типа вакцины предпочтительна та или иная версия мРНК); одна мРНК, кодирующая только рецептор-связывающий домен (RBD) S-гликопротеина, т.к. антитела, направленные прицельно против RBD, могут быть более эффективными и в случае развития инфекции смогут обеспечивать ее более легкое течение; и антитела, соответствующие самоамплифицирующейся мРНК (поскольку этот формат требует гораздо более низких доз, чем нереплицирующаяся мРНК) [20]. Для клинических исследований компания BioNTech решила выбрать вакцину, которая включала мРНК, кодирующую полноразмерный S-гликопротеин в «prefusion» (см. далее) конформации (BNT162b2), первая инъекция которой была сделана 23 апреля 2020 г., а результаты I фазы данной вакцины в дозах 10 мкг, 30 и 100 мкг продемонстрировали сероконверсию у всех добровольцев и высокие титры нейтрализующих антител, при этом показатели были аналогичными или превышали таковые у пациентов, перенесших COVID-19, включая участников старше 65 лет. Кроме того, вакцина показала хороший профиль безопасности [21]. Объявив о своем сотрудничестве 17 марта 2020 г., Pfizer и BioNTech инициировали совместное исследование вакцины, что значительно ускорило набор участников. Исследование фазы III было завершено уже 18 ноября 2020 г. В нем приняли участие более 40 тыс. участников и была продемонстрирована 95%-ная эффективность в предупреждении COVID-19. Связанные с вакциной побочные эффекты наблюдались у 21% участников в группе вакцины и 5% в группе плацебо. Основываясь на этих благоприятных данных по безопасности и эффективности, BNT162b2 стал первым когда-либо одобренным препаратом на основе мРНК и первой разрешенной вакциной против COVID-19. Первую вакцину для экстренного использования одобрила Великобритания 2 декабря 2020 г., а 11 декабря 2020 г. – США; 31 марта 2021 г. Pfizer и BioNTech сообщили о 100%-ной эффективности вакцины для подростков в возрасте 12–15 лет по результатам проведенного клинического исследования 3-й фазы, включившего 2260 участников [22]. Первого апреля 2021 г. компания Pfizer сообщила о том, что после 6-месячного периода отслеживания вакцинированных пациентов и 927 подтвержденных симптоматических случаев COVID-19 эффективность вакцины составила 91,3%, при этом данный показатель для предупреждения тяжелых случаев инфекции достиг 100%, а также показал эффективность на уровне 100% для предупреждения случаев инфекции SARS-CoV-2, вызванных проблемным вариантом вируса В.1.351 [23].
На 1 апреля 2021 г. мРНК вакцина Pfizer/BioNTech получила разрешение на использование в Бахрейне, Бразилии, Новой Зеландии, Саудовской Аравии, Швейцарии, а также на применение по экстренным показаниям в Аргентине, Австралии, Ботсване, Канаде, Чили, Колумбии, Коста-Рике, Европейском Союзе, Исландии, Израиле, Японии, Великобритании, США и в ряде других стран [24].
Структурные особенности молекулы мРНК
Итак, что же сделали разработчики вакцины с исходной молекулой мРНК, отвечающей за синтез S-гликопротеина? Прежде всего, если мы посмотрим даже на первые 500 символов кода (рис. 2), то можно сразу отметить, что вместо привычного для молекулы РНК уридина (U) включен псевдоуридин (Ψ). Этот исключительно важный этап преобразования молекулы мРНК представляет собой относительно давно разработанный подход и его применение не вызывает удивления [25, 26]. Эксперименты показали, что мРНК, в которых уридин заменен на псевдоуридин, хуже распознаются иммунной системой, что, по всей видимости, связано с работой клеточных систем по идентификации собственных и вирусных РНК, что было озвучено, в частности, в работе K. Kariko et al. [27]. Более того, вакцина на основе мРНК от Pfizer/BioNTech содержит не чистый псевдоуридин, а метил-псевдоуридин (1-метил-3’-псевдоуридин или m1Ψ). В ряде исследований показано, что данная модификация псевдоуридина улучшает синтез белка при использовании мРНК [28]. Не следует думать, что псевдоуридин по своей сути – измененное или чужеродное соединение, т.к. он встречается во многих типах РНК у разных организмов, в т.ч. и человека.

Кроме того, вполне вероятно, что в связи с имеющимися различиями в составах кодонов мРНК вирусов летучих мышей и человека [29] разработчики Pfizer/BioNTech также заменили соответствующие кодоны «человеческими», которые, естественно, более узнаваемы иммунной системой и не инициируют процесса разрушения мРНК.
Cap или Five-prime cap (5’Сap), m7G+m3’-5’-ppp-5’-Am, Позиции 1–2. 5’Сap (от англ. Cap – кепка, шляпа) представляет собой начальный и исключительно важный участок мРНК, своего рода «пропуск» к процедуре трансляции, без которого мРНК будет распознана как чужеродная и уничтожена. 5’Сap выполняет ряд функций [30, 31], которые включают регуляцию ядерного экспорта, предупреждение деградации нуклеазами, промоцию трансляции и др. Важно, что 5’Сap может устранять свободные фосфатные группы в последовательности мРНК, что значительно повышает стабильность мРНК и позволяет рибосоме распознавать начало мРНК, повышает эффективность трансляции за счет связывания с эукариотическим фактором инициации трансляции 4E (eIF4E) [32, 33]. Очевидно, что модификация 5’Cap может иметь решающее значение для улучшения свойств мРНК. Существует два общих подхода к кэпированию мРНК in vitro. Первый вариант – добавление обычного Сар-аналога, структуры m7GpppG, в систему транскрипции мРНК может обеспечить кэпирование мРНК наряду с транскрипцией in vitro; второй – путем кэпирующей ферментативной реакции после начальной транскрипции in vitro [30, 32]. Вполне очевидно, что различные варианты кэпирования обусловливают различную эффективность трансляции, в связи с чем разработчики Pfizer/BioNTech подобрали такую структуру 5’Сap, которая бы обеспечила оптимальный, а возможно максимальный, уровень трансляции.
5’-не транслируемый участок (5-prime untranslated region, 5’-UTR), Позиции 3–54. С данного конца (5’-UTR) рибосома начинает процедуру синтеза белка, считывая информацию с мРНК. Исключение последовательности гена в 5’-UTR, идентичной начальной последовательности ORF, может эффективно предотвращать ложный старт и изменение рамки считывания во время процесса трансляции мРНК [34]. Кроме того, некоторые конкретные последовательности могут быть добавлены к 5’-UTR для повышения стабильности мРНК и повышения точности трансляции. В частности, M. Kozak et al., включив последовательность GCC-(A/G)-CCAUGG в этой области, показали, что это приводит к более точному запуску процесса трансляции [35]. Исследования также показывают, что высокостабилизированная вторичная структура 5’-UTR будет препятствовать связыванию рибосом с мРНК, а короткие и слабосвязанные 5’-UTR более подходят для процессов трансляции [36].
Что же привнесли Pfizer/BioNTech в качестве структурной особенности 5’-UTR в молекуле мРНК в составе вакцины? В UTR помимо прочего содержатся мета-данные, указывающие на то, когда должна начаться трансляция и в каком объеме будет осуществлен синтез [37]. Разработчики обнаружили наиболее «ранний» из возможных UTR, инструкции в отношении которого были взяты из гена α-глобина. Известно, что данный ген обусловливает стабильное производство массы белка, чем, в частности, объясняется длительный период жизни эритроцитов при постоянной работе линии по синтезу протеинов. Кроме того, показано, что участки 5’-UTR мРНК α-глобинов приводят к более высокому уровню трансляции по сравнению с β-глобинами [38].
Открытая рамка считывания (ORF – Open Reading Frame). Начинаясь с сигнального пептида, является кодирующей, а следовательно, ключевой областью мРНК. Скорость трансляции области ORF исключительно критична, поэтому выбор подходящих кодонов в этой области может оптимизировать эффективность трансляции мРНК в целом. Оптимизированная последовательность ORF обычно включает синонимичные частые кодоны и/или кодоны с более высоким содержанием тРНК для замены редких кодонов таким образом, что гены с высокой экспрессией могут транслироваться с использованием тех же кодонов хозяина [39]. Однако высокая скорость трансляции мРНК не всегда выгодна, поскольку некоторым белкам требуется низкая скорость трансляции для правильного и эффективного фолдинга; в данном случае использование кодонов с низкой частотой в ORF может обеспечить белковые продукты более высокого качества [39, 40]. Следовательно, для разных антигенов должны использоваться разные стратегии оптимизации, чтобы улучшить скорость трансляции мРНК и одновременно гарантировать качество экспрессируемого антигена.
Сигнальный пептид S-гликопротеина (S-glycoprotein signal peptide), позиции 55–102. Ключевой функцией сигнального пептида S-гликопротеина является направление транслокации растущей полипептидной цепи в эндоплазматический ретикулум, поэтому эта последовательность еще называется «лидерной», т.к. определяет путь синтезированного белка с конкретной мРНК [41]. Сигнальная последовательность не очень длинная, однако на примере кода на рис. 3 видно различие между РНК вируса и вакцины (для удобства сравнения Ψ заменен на U).

На что здесь следует обратить внимание прежде всего и что важно именно для разработки вакцины? Модификации вакцины (UUU → UUC) синонимичны, т.е. при коде, отличном от оригинального, мы получаем продукцию аналогичных белков. Бόльшая часть изменений содержится в третьем кодоне, при этом, как уже было сказано, это синонимичные изменения, но с какой тогда целью они были сделаны? Достаточно четко можно увидеть, что почти все модификации увеличивают количество C и G, и, как было показано в экспериментах, именно РНК с большим количеством G и C значимо активирует процесс синтеза протеинов [8, 42]. Именно поэтому разработчики Pfizer/BioNTech там, где это было возможно, сделали замены на G и C.
Спайк-гликопротеин (Spike (S)-glycoprotein), позиции 103–3879. Как видно из рис. 1, строго говоря, данная последовательность носит название S_protein_mut, т.к. в ней также (как и в случае модификаций с G и С) применена процедура оптимизации кодонов, в частности, за счет мутаций K986P и V987P (3874-3879), результатом которых является замена лизина (К) и валина (V) на пролин (Р) в указанных позициях, что обусловливает фиксацию S-гликопротеина в «открытой» конформации – т.е. антиген-оптимальной конформации, при которой он способен соединяться с АПФ2-рецепторами клеток. В 2017 г. было описано, что двойная замена пролина (S-2P) в конкретных локусах заставляет белки SARS-CoV-1 и MERS принимать данную конформацию даже без присоединения к вирусу, что происходит, видимо, благодаря прочности самого пролина [43, 44]. Это несомненно важное преобразование, т.к. нам необходим синтез антител именно к «рабочей» открытой конформации S-гликопротеина. Справедливости ради надо отметить, что к данной хитрости прибегли и разработчики мРНК вакцины компании Moderna [45].
В конце белка находится стоп-кодон, помеченный буквой «s». Это указывает на окончание белка. У вируса в качестве стоп-кодона выступает UAA, в вакцине Pfizer/BioNTech – два кодона UGA. Точная причина этого не ясна.
3’-не транслируемый участок (3-prime untranslated region, 3’-UTR), позиции 3880–4174. 3’-UTR – по всей видимости, наименее изученный в функциональном плане участок мРНК, однако известно, что 3’-UTR может принимать участие в регуляции эффективности трансляции, стабильности мРНК, содержать сигналы полиаденилирования и выполнять ряд других регуляторных функций [46, 47]. Интересно, что 3’-UTR в целом считается концентрированной областью нестабильных факторов, поэтому их минимизация, а равно как введение стабильных элементов, могут значимо повышать стабильность мРНК и увеличивать период ее жизни [48, 49]. Вполне логично, что особенностью 3’-UTR мРНК в вакцине от Pfizer/BioNTech является как раз отсутствие т.н. AU-богатых участков (AU-rich elements), т.е. участков, состоящих преимущественно из нуклеотидов А и U, которые служат сигналом для привлечения ферментов, приводящих к быстрой деградации мРНК [50]. Убрав данные нуклеотиды, разработчики Pfizer/BioNTech значительно увеличили стабильность мРНК в составе вакцины.
Поли(А)-«хвост» (Poly(A)-tail), позиции 4175–4284. Поли(A)-«хвост», как и структуры 5’-UTR, является ключевым элементом во время трансляции мРНК. Последовательность поли(A) может замедлять процесс деградации РНК экзонуклеазами, что увеличивает стабильность, продлевает период полужизни in vivo и повышает эффективность трансляции мРНК. Ранние исследования показали, что длинная последовательность поли(A) может улучшать стабильность мРНК. Например, оптимальная длина последовательности поли(A) в дендритных клетках составляет примерно 120–150 нуклеотидов [32, 51], а увеличение длины последовательности поли(A) до более чем 300 нуклеотидов в Т-клетках человека может стать более благоприятной для увеличения стабильности и трансляционной эффективности мРНК [52]. Когда длина последовательности поли(A) меньше 20 нуклеотидов, это снижает эффективность трансляции мРНК [53], т.к. мРНК может использоваться несколько раз, по мере ее участия в процессе синтеза белка она теряет остатки А и «хвост», по сути, укорачивается. Так как он является местом прикрепления защитных белков, не позволяющих приводить к деградации мРНК, исчезновение А и укорочение поли(А)-«хвоста» в целом в конечном итоге приводят к разрушению молекулы. В связи с этим вполне очевидно, что чем длиннее поли(А)-хвост, тем больше будет период жизни мРНК [54]. Данная молекула мРНК заканчивается 110-нуклеотидным поли(А)-«хвостом», который содержит 30 остатков аденозина (аденозин монофосфат), прерывающихся 10-нуклеотидной связывающей последовательностью, за которой следуют еще 70 остатков аденозина. Это действительно длинная последовательность, что также входило в цели разработки вакцины Pfizer/BioNTech (рис. 4).
Заключение
В заключение необходимо сказать, что появление первых вакцин на основе мРНК стало большим шагом вперед не только в разработке вакцин против SARS-CoV-2, но и в вакцинопрофилактике в целом. Доказанные в клинических исследованиях и подтвержденные в реальной практике эффективность и безопасность, по сути, обозначили потенциальную возможность применения и значимость технологии на основе молекул мРНК при создании современных вакцин. Крайне важны разработка самой молекулы мРНК, точнее ее модификация, и выбор наиболее подходящего для конкретной ситуации варианта.
Несмотря на свою простую структуру, мРНК вакцины можно оптимизировать многими способами и подходы к этой оптимизации начали изучаться около 20 лет назад. Примером такой оптимизации может быть композиция вакцины Pfizer/BioNTech, которая представляет собой отнюдь не просто копию вирусной мРНК, ответственной за синтез S-гликопротеина вируса, а действительно сложный биотехнологический продукт, в котором ряд превращений позволил обеспечить стабильность молекулы мРНК, увеличив продолжительность ее жизни и оптимизировав сам процесс синтеза белка.
Необходимо понимать, что с момента открытия С. Бреннером мРНК в 1961 г. [55] до разработки самой концепции вакцин на основе мРНК в 1990 г. Дж. Вульфом [56] и тем более до регистрации первых вакцин потребовался огромный объем научных наработок, отбор кандидатов и проведение исследований на человеке. Несмотря на свою относительно короткую «историю», не вызывает сомнений, что технологии использования мРНК ни в коем случае не останутся актуальными только для решения вопроса профилактики COVID-19, но будут активно развиваться для решения ряда задач в других областях медицины.


