Актуальность
Проблема устойчивости микроорганизмов к противомикробным препаратам (ПМП), в частности штаммов с множественной лекарственной устойчивостью (МЛУ) и широкой лекарственной устойчивостью (ШЛУ), представляет первостепенную угрозу для здравоохранения во всем мире, вызывая около 1,3 млн летальных исходов ежегодно. По данным Всемирной организации здравоохранения (ВОЗ), повышенная устойчивость к ПМП входит в тройку основных проблем общественного здравоохранения в мире в XXI в. При этом наибольшую опасность в популяции представляет группа микроорганизмов под аббревиатурой ESKAPE (Enterococcus, Staphylococcus, Klebsiella, Actinobacter, Pseudomonas и Enterobacter) [1–2].
Данная тенденция обусловлена неадекватным (примененные резервных ПМП, нарушение режимов кратности, дозирования, продолжительности лечения), повсеместным (в виде профилактических средств) использованием ПМП в медицинской, сельскохозяйственной и ветеринарной отраслях, а также отсутствием новых ПМП на рынке, обусловленным высокими экономическими затратами на синтез с колоссальными бюрократическими барьерами [3–11].
Совокупность вышеописанных факторов приводит к возможности микроорганизмов развивать свои молекулярные механизмы адаптации, направленные на увеличение внутривидовой выживаемости, факторов агрессии и инвазии, таких как производство инактивирующих ферментов (например, β-лактамазы расширенного спектра, фосфотрансферразы, ацетилтранферразы и др.), изменение мишеней для антибиотика (например, метилирование остатка аденина, в случае устойчивости к эритромицину), изменение структурной архитектоники клеток (например, мутация гена порина у Гр-отрицательных бактерий), эффлюкс ПМП (выдавливание препарата за пределы клетки путем активации «насосов» для оттока), активация альтернативных путей метаболизма (например выработка альтернативного пути синтеза фолиевой кислоты при воздействии сульфаниламидов), транспорт генов устойчивости внутри колоний бактериальных клеток, способность к формированию клеток-персистеров (метаболически не активные клетки с низкой чувствительностью ПМП), а также образование биополимерных матриц с электростатическим компонентом, препятствующее проникновению ПМП в бактериальную колонию [12–17].
Вышеописанное диктует необходимость поиска новых подходов к лечению заболеваний, инициированных штаммами с МЛУ и ШЛУ, целью которых являются уменьшение смертности, спровоцированной вышеописанной микрофлорой, снижение затрат на здравоохранение, повышение эффективности лечения и сокращение сроков реабилитации пациентов с различной инфекционной патологией.
В последние годы, одним из перспективных направлений в данной области является исследование антиинфекционной активности наночастиц, которые объединяют под общим термином «антибактериальная нанотерапия, или наноантибиотики». Наночастицы представляют собой синтезированные, как правило химическим способом, инженерные структуры сверхмалого размера 1–100 нм, имеющие различную форму, состав, биологические и физико-химические свойства, которые определяют спектр их антимикробной активности, которая также в значительной степени может зависеть от температуры, pH, концентрации ионов металлов, особенностей функционализации, силанизации и других биологических факторов [18–21].
Существующее разнообразие наночастиц широко представлено липосомами, мицеллярными структурами, твердыми липидными наночастицами, ноногелями, нанокапсулами, нанотрубками, эмульсиями, а также полимерными, углеродными, металлическими наноточками (КТ) [22–23].
Наиболее перспективными и изученными являются металлические наночастицы в силу простоты и предсказуемости их производства, безо-пасности, химической и физической стабильности, моделируемости их оптических и электронных свойств. Возможность их изучения посредством УФ-спектроскопии, масс-спектрометрии, рентген-структурного анализа, а также различных видов электронной микроскопии определяет потенциальные перспективы для поиска механизмов воздействия КТ на возбудителей инфекции [24–26].
Потенциальные механизмы антиинфекционной активности КТ определяются их способностью к проникновению внутрь бактериальной клетки за счет сверхмалого размера (3–5 нм) и ее разрушению за счет дозированного производства активных форм кислорода (АФК), сопряженных со свободными электронами парами на внешнем энергетическом уровне КТ, а также способность инициировать окислительный стресс внутри бактерии с поражением белков, ферментов, ядерного аппарата, насосных помп и других жизненно важных элементов. Электростатическая адсорбция КТ на клеточной стенки бактерии приводит к деполяризации мембран, снижению ее проницаемости и текучести, приводящей к гибели клетки. Взаимодействуя с отрицательными зарядами поверхности бактерии (через карбоксильные и фосфатные группы), положительно заряженные ионы КТ также оказывают бактерицидное действие [27–30].
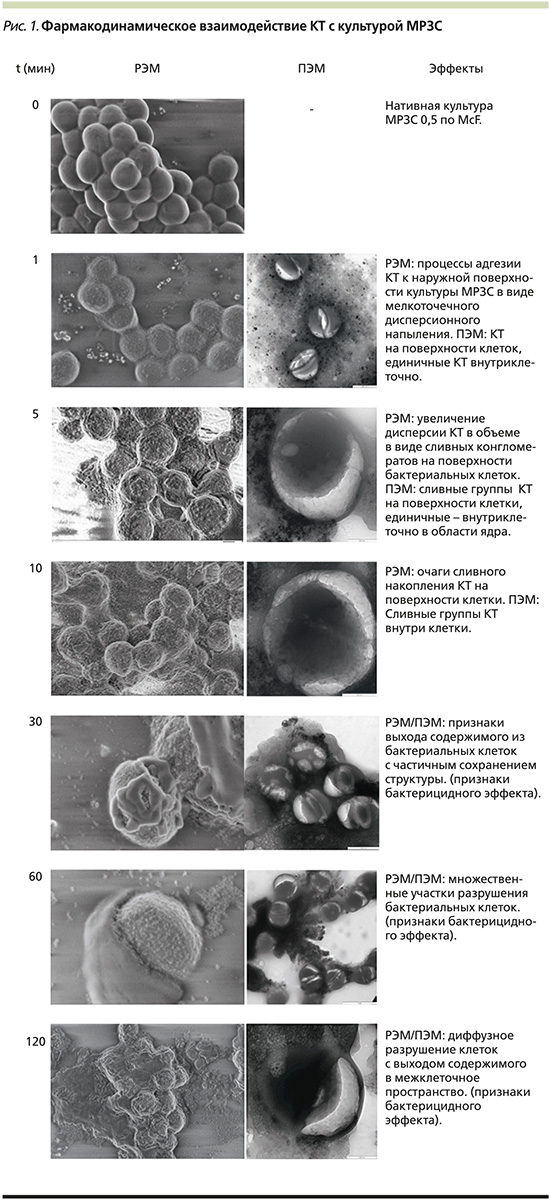
В качестве средств детектирования нанаоразмерных объектов, описания механизмов их потенциального бактерицидного действия, а также отображения наноструктурных и клеточных взаимодействий целесообразно использовать современные микроскопы субмикронного разрешения – растровую/просвечивающую электронную микроскопию, которую применяют для изучения основ физико-биологического взаимодействия живых и неживых систем.
В частности, детектирование наноразмерных частиц во время их взаимодействия с бактериальной клеткой может демонстрировать как признаки бактерицидного эффекта – набухание клетки, выход ее содержимого в межклеточное пространство, изменение конфигурации и расположения клеточных органелл, признаки разрушения клеточной стенки, так и признаки отсутствия взаимодействия с наночастицами – взаимоотталкивание, образование конгломератов вне клетки, отсутствие в поле зрения.
На основании вышеизложенного особую актуальность приобретает необходимость исследования динамических закономерностей взаимодействия металлических наночастиц непосредственно с бактериальной клеткой, а также вопросов, связанных с предполагаемыми фармакологическими эффектами, оказываемыми ими, что послужило целью данного исследования.
Цель исследования: исследование фармакодинамики металлических наночастиц при взаимодействии с бактериальной клеткой для определения потенциальной перспективы лечения резистентной бактериальной инфекции.
Методы
Исследование проводилось на базе университетского научно-образовательного центра «Наноматериалы и нанотехнологии» Уральского федерального университета (УРФУ) им. первого президента России Б.Н. Ельцина и лаборатории электронной микроскопии сверхвысокого разрешения УРФУ, Екатеринбург.
В качестве метода исследования особенностей фармакодинамики КТ при взаимодействии с бактериальной клеткой применялась растровая (РЭМ) и просвечивающая электронная микроскопии (ПЭМ).
В качестве РЭМ использовался электронный микроскоп Sigma VP, Carl Zeiss (Германия) в режиме высокого вакуума. Исследуемые образцы наносились на углеродную ленту перед помещением в камеру микроскопа.
В качестве ПЭМ использовался микроскоп JEM-2100 (Япония). Во всех случаях перед исследованиями на образцы наносилась аморфная углеродная пленка. Образцы закреплялись на держателях и также помещались в камеру электронного микроскопа.
В качестве объекта исследования брался водный раствор металлических КТ InP/ZnSe/ZnS 0.1 мл в концентрации 0,001% (разведение для наилучшей визулизации получено путем титрования водного раствора) и культура митициллин-резистентного золотистого стафилоккка (МРЗС) в мутности 0,5 по стандарту МакФарланда (McF) путем ее взятия непосредственно с кровяного агара в объеме 0,1 мл. Зона задержки роста (ЗЗР) у исследуемого штамма (n=12, n – кратность исследований) к Ванкомицину/Левофлоксацину составляла в среднем 12±0,5/6±0,5 мм соответственно.
Культура исследовалась методом РЭМ/ПЭМ в чистом виде, а также после смешивания с раствором КТ в равных пропорциях во временных интервалах через 1, 5, 10, 30, 60 и 120 минут соответственно для оценки особенностей фармакодинамики.
В качестве метода контроля оценки антиинфекционной активности КТ применялся диско-диффузионный метод.
Результаты
Результаты РЭМ/ПЭМ, отображающие взаимодействие КТ с культурой МРЗС, представлены на рис. 1, из которого видно, что данные по кинетике процесса РЭМ коррелируют с данными, полученными в результате проведения РЭМ. Этапы адгезии КТ на поверхности бактериальной клетки начинаются через 5 минут после их взаимодействия и, вероятно, обусловлены электростатическими процессами; КТ свободно проникают через клеточную мембрану бактериальной клетки (в частности, МРЗС); первые признаки разрушения бактериальной клетки с выходом ее содержимого начинают визуализироваться через 30 минут наблюдения, более того, вовлечение ядерного аппарата в процесс ее разрушения фиксируется практически на всех этапах наблюдения; последующая динамика разрушения сопровождается генерализованным выходом содержимого бактериальных клеток в межклеточное пространство, изменением их формы и объема в течение 60–120 минут, что указывает на бактерицидный эффект.
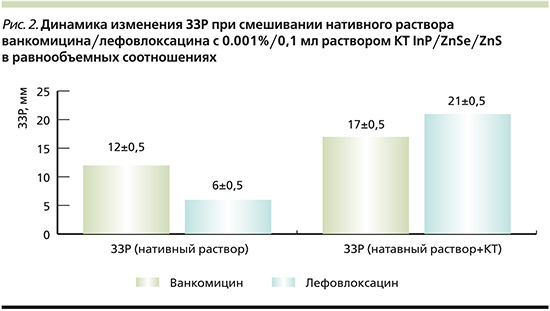
Динамика изменения ЗЗР при смешивании нативного раствора Ванкомицина/Лефовлоксацина с 0,001%/0,1 мл раствором КТ InP/ZnSe/ZnS в равнообъемных соотношениях (n=12, где n – число разведений) отображена на рис. 2, из которого видно увеличение ЗЗР Ванкомицина/Левофлоксацина на 5 и 15 мм соответственно при добавлении к ним раствора КТ, что свидетельствует о значительном увеличении антиинфекционной активности исследуемых АБ в отношении данного штамма МРЗС.
Обсуждение
Общеизвестно, что появление бактерий с МЛУ происходит во всем мире, что ставит под угрозу эффективность антибиотиков, которые кардинально изменили клинические подходы и спасли миллионы жизней [33, 34].
Кризис устойчивости к антибиотикам объясняется чрезмерным и неправильным использованием этих препаратов, а также отсутствием разработки новых лекарств фармацевтической промышленностью из-за снижения экономических стимулов и жестких нормативных требований [35–37].
На сегодняшний день крайне необходимы скоординированные усилия для реализации новой политики, возобновления исследований и принятия мер по преодолению кризиса антибиотикорезистентности, уменьшения опосредованной смертности, инвалидности и снижения затрат на здравоохранение, что послужило стимулом для проведения данного исследования.
На основании экспериментов с проведением РЭМ и ПЭМ было показано, что КТ обладают потенциалом к проникновению непосредственно во внутриклеточное пространство через сформированные мембранные каналы. Механизмы, происходящие в клетке после проникновения КТ, сопровождаются ее полным разрушением, предположительно физико-химической основой которых является процессы, описанные выше, основанные на работах [27–30], что является проявлением бактерицидного эффекта КТ.
Данные, демонстрирующие увеличение ЗЗР Ванкомицина/Левофлоксацина на 5 и 15 мм соответственно, при добавлении к ним раствора КТ, свидетельствуют о значительном увеличении антиинфекционной активности исследуемых антибиотиков в сочетании с КТ в отношении данного штамма МРЗС.
На основании данных наблюдений стартом антиинфекционной активности КТ в отношении патогенной микрофлоры можно рассматривать временной промежуток от 30 до 60 минут, к 120 минутам наблюдения практически все бактериальные клетки находящиеся в зоне наблюдения,полностью или частично разрушены.
Актуальность данной работы состоит в том, что впервые определены поэтапные (фармакодинамические) характеристики механизма взаимодействия металлических полупроводниковых КТ с бактериальной клеткой МРЗС, описаны процессы взаимодействия с клеточной мембраной и основными клеточными органеллами. Установлена взаимосвязь между проникновением КТ внутрь клетки и выходом ее содержимого в межклеточное пространство.
Получены данные о значительном увеличении ЗЗР у исследуемых антибиотиков при смешивании их с исследуемыми КТ.
Ограничением данного исследования являются малый объем видов клеточных культур и типов КТ, используемых в исследовании, отсутствие обоснованных научных данных о химических процессах, происходящих внутри бактериальной клетки при взаимодействии с конкретным типом КТ, отсутствие данных по минимально ингибирующей концентрации исследуемых КТ в отношении изучаемого возбудителя инфекций.
Заключение
Полученные результаты демонстрируют перспективность дальнейших исследований, направленных на изучение технологии сочетанного (конъюгированного) использования КТ с актуальными антиинфекционными агентами, для повышения их антиинфекционной активности, снижения риска селекции штаммов с МЛУ и перспективой снижения затрат на здравоохранение.
Вклад авторов. Концепция и дизайн исследования – В.О. Пономарев, В.В. Омельяновский. Сбор и обработка материала – В.О. Пономарев. Статистическая обработка данных – В.О. Пономарев. Написание текста – В.О. Пономарев, В.В. Омельяновский. Редактирование – В.В. Омельяновский.
Благодарности. Авторы выражают благодарность сотрудникам научно-образовательного центра «Наноматериалы и нанотехнологии» Уральского федерального университета (УРФУ) им. первого президента России Б.Н. Ельцина и сотрудникам лаборатории электронной микроскопии сверхвысокого разрешения УРФУ, Екатеринбург, в частности доценту Вохминцеву Александру Сергеевичу и профессору Вайнштейну Илье Александровичу, за помощь в организации исследования.



